tslearn使用輪廓係數(silhouette_score)評估KShape聚類效果
前言
tslearn和sklearn一樣,是一款優秀的機器學習框架,tslearn更偏向於處理時間序列問題,如其聚類模組就包含了DTW(Dynamic Time Warping)等演算法及變種,也提供了輪廓係數對聚類效果評估,十分方便。但可惜,tslearn似乎沒有提供對KShape聚類的評估方法,而且tslearn用的人也不多,官方文件也是很 「簡潔」,網上也搜不到多少相關文章,所以這裏也就記錄下自己的踩坑過程
輪廓係數評估介面呼叫
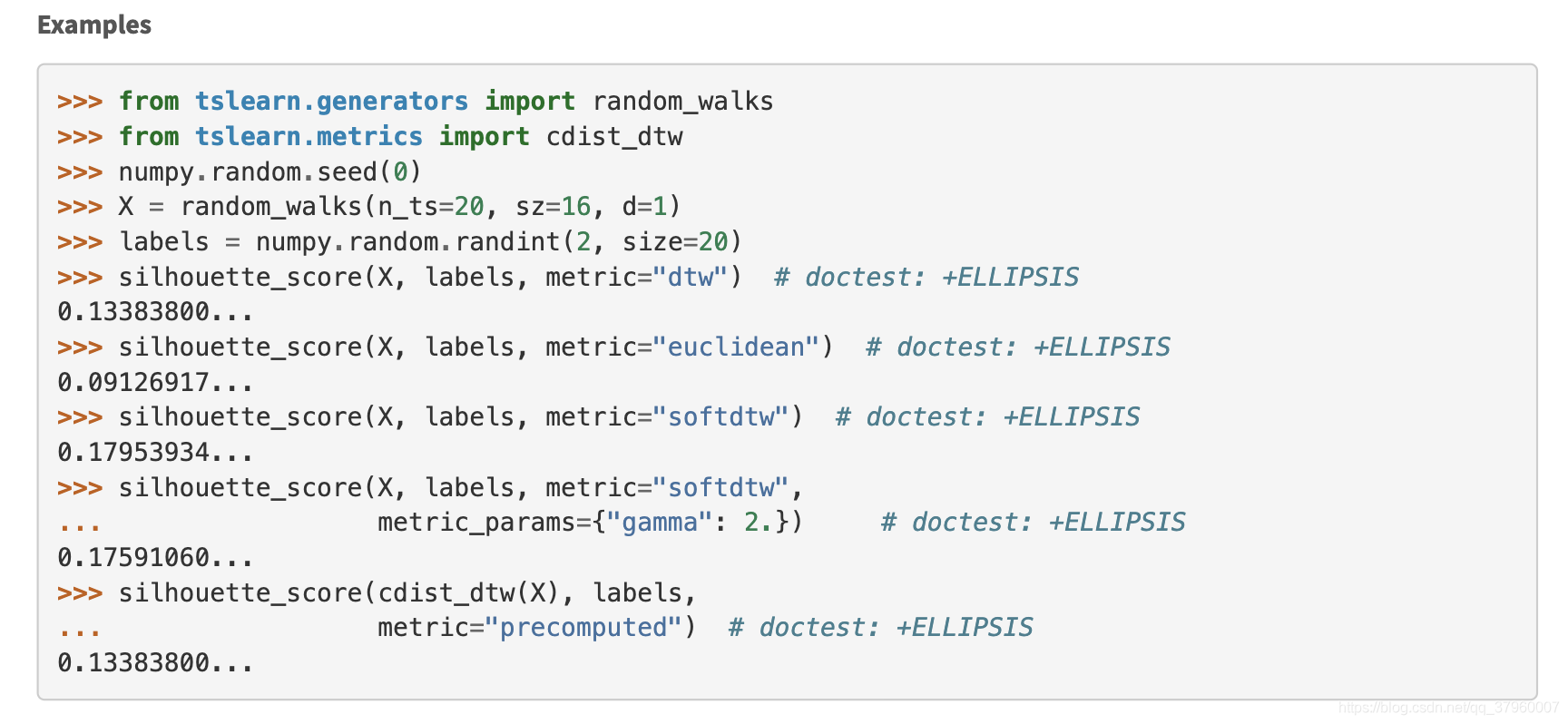
先看官方例子,這裏X是一個三維的numpy陣列,代表20段時間序列,每段序列16個時間點。labels代表每段時間序列(每條時間曲線)的聚類結果,metric是每條時間曲線之間相似度度量方法,可以看到官方提供了dtw-dba、softdtw以及歐氏距離三種相似度度量方法,但沒有關於KShape聚類的。
注意到最後一行,metric=「precomputed」,說明官方提供了使用者自定義的距離度量方法,很好,那麼使用silhouette_score評估KShape的關鍵就在此了。又注意到precomputed的時候,參數傳的是cdist_dtw(X),而其他傳的是X,有什麼區別?
列印
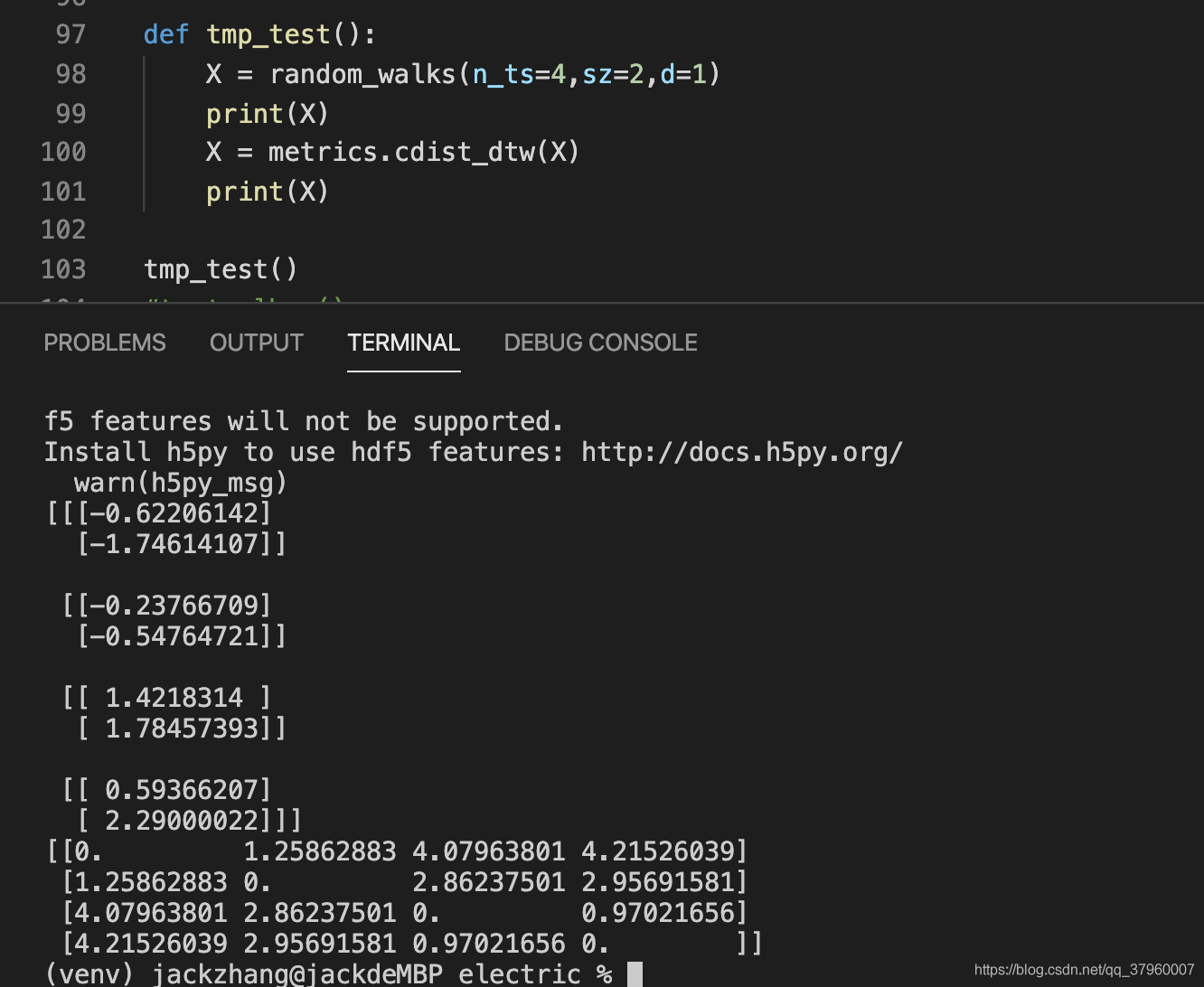
醒目的零對角線告訴我們這是X與X自身的距離矩陣,相同時間曲線的距離爲零。這樣其實也就對了,輪廓係數的計算本來就需要比較當前元素到同簇下其他元素的距離,以及與其他簇下的距離,因此需要得到每個元素之間的距離,X與X自身的距離矩陣恰好解決了這點,而計算其他簇就需要labels標籤指明哪些元素屬於哪些類
問題來了,那我怎麼知道KShape聚類用什麼方法度量相似度?不清楚其用的方法,就得不到距離矩陣,也就無法使用precomputed。自己看論文仿寫一個?仿寫的若與原始碼的實現不同,細節上的差距也可能會導致輪廓係數的評估不準 不準
原始碼找突破口
我們現在想要的是KShape對時間序列相似度、也就是距離的計算方法,而且要原封不動,不能仿寫,所以只能去KShape原始碼裡尋找結果。點進KShape,大概是一千多行的位置
既然是距離,就在KShape行數往後搜尋dist,看看能否找到相關線索
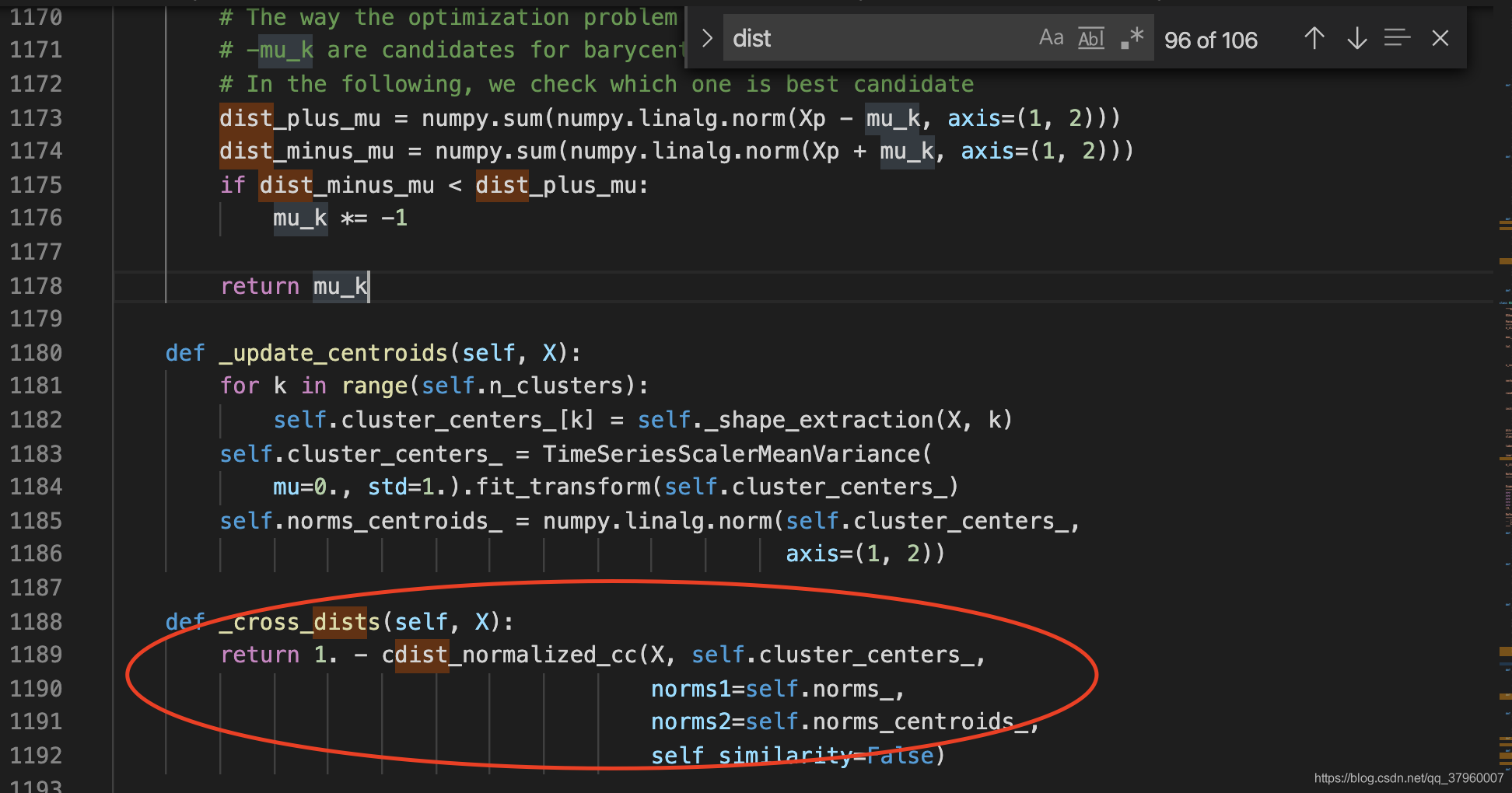
一個醒目的函數名引起了我的注意,cross_dist,交叉距離,似乎有點距離矩陣的味道。於是我查閱了KShape官方文件,看到有這麼一條參照:

說明tslearn裡的KShape原始碼是按該論文實現的。在論文k-Shape: Efficient and Accurate Clustering of Time Series中,看到了這樣一個公式:

表達式 1 - max(xxx),這不正好和_cross_dist函數的結構一樣?就差一個max。
繼續點進 cdist_normalized_cc 函數,發現點不進去,又去import的地方繼續點,發現 tslearn.cycc 也點不進去
最後發現cycc是 .so檔案
既然是呼叫的其他拓展語言的庫,就只能去github上看了。找到官方的github後點開cycc.pyx檔案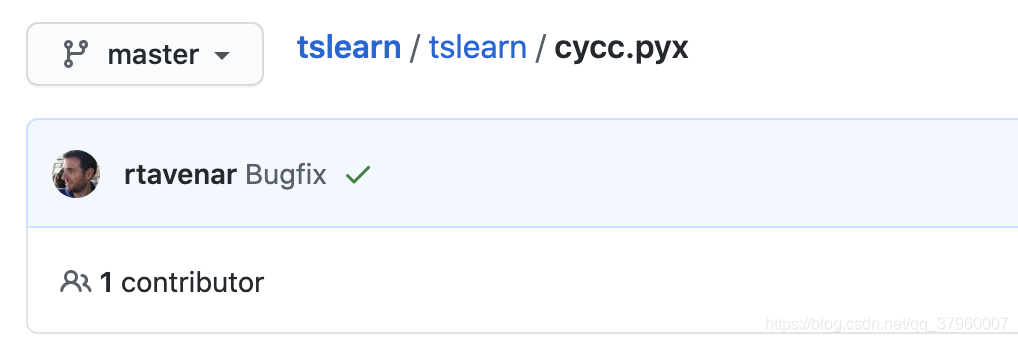
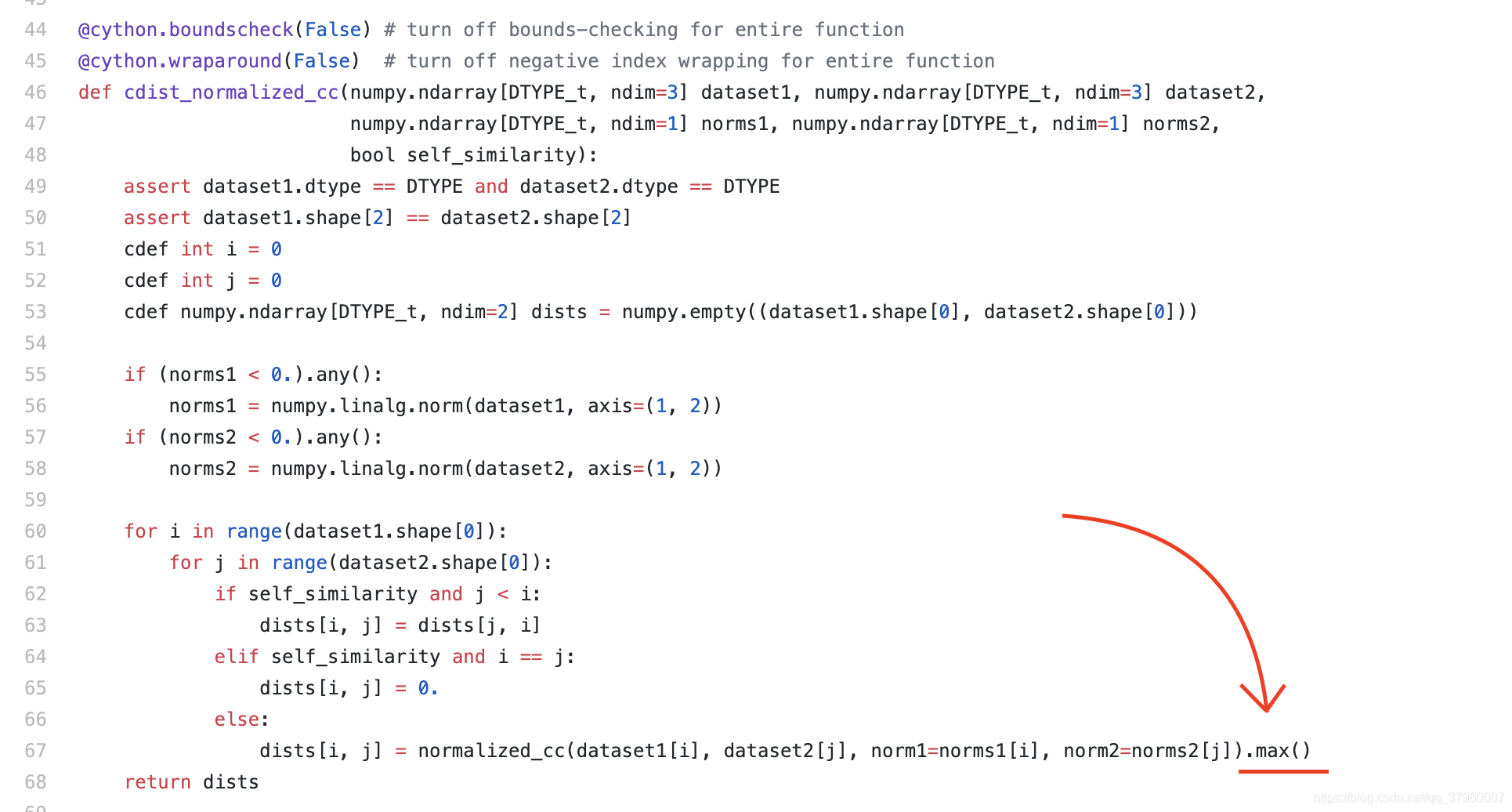
在 cdist_normalized_cc 函數的最後有一個max!這不正好完全與論文中的公式對應了?也就是說KShape聚類採用的是一個叫SBD的方法進行距離計算的,其實現就在github的 cycc.pyx檔案中,也就是說我們只需拿到這段程式碼就可以生成X的距離矩陣,從而可以使用precomputed進行輪廓係數對KShape的聚類效果評估
參數確定
可以看到 cdist_normalized_cc 有五個參數,dataset1和dataset2代表計算兩者之間的距離矩陣,那麼這兩個參數都填 X 即可。還有兩個norms1和norms2參數,看論文裡的公式,向量平方後開根號,似乎在求向量的模長。實驗一下,直接修改原始碼,列印norms的值
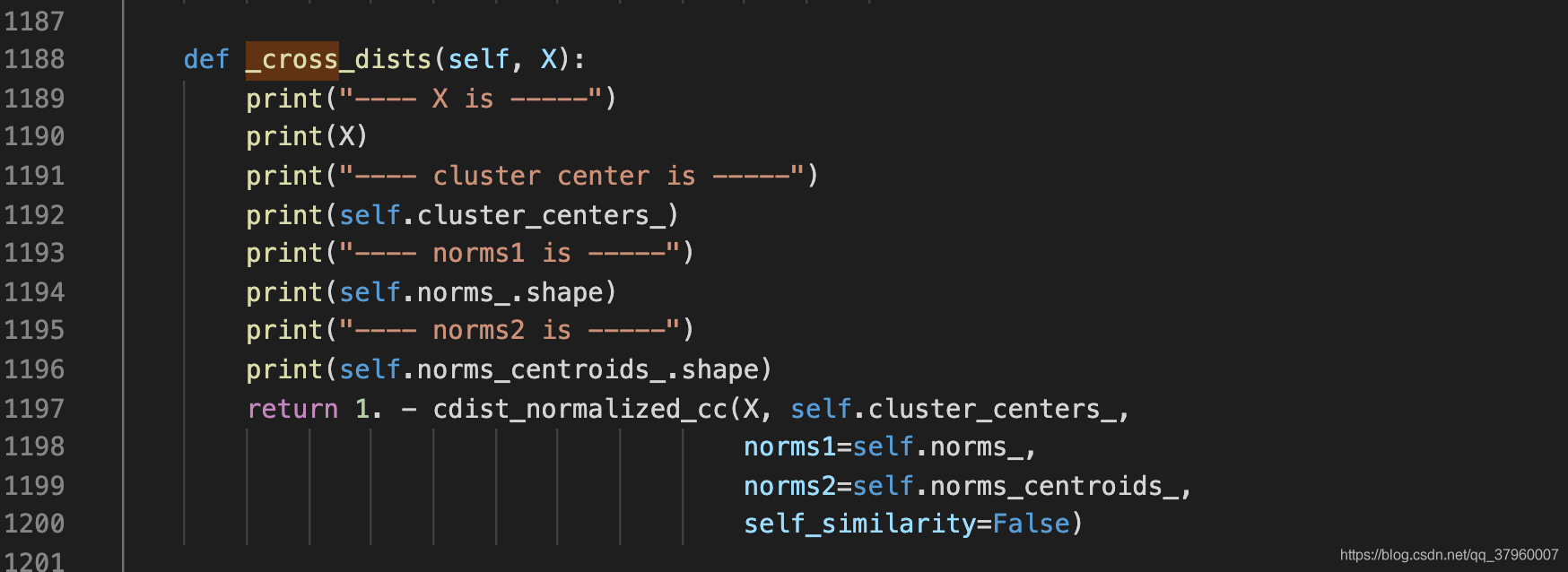
按照官方的案例,random_walk生成X和labels、初始化KShape並呼叫 fit 函數後,結果如下:
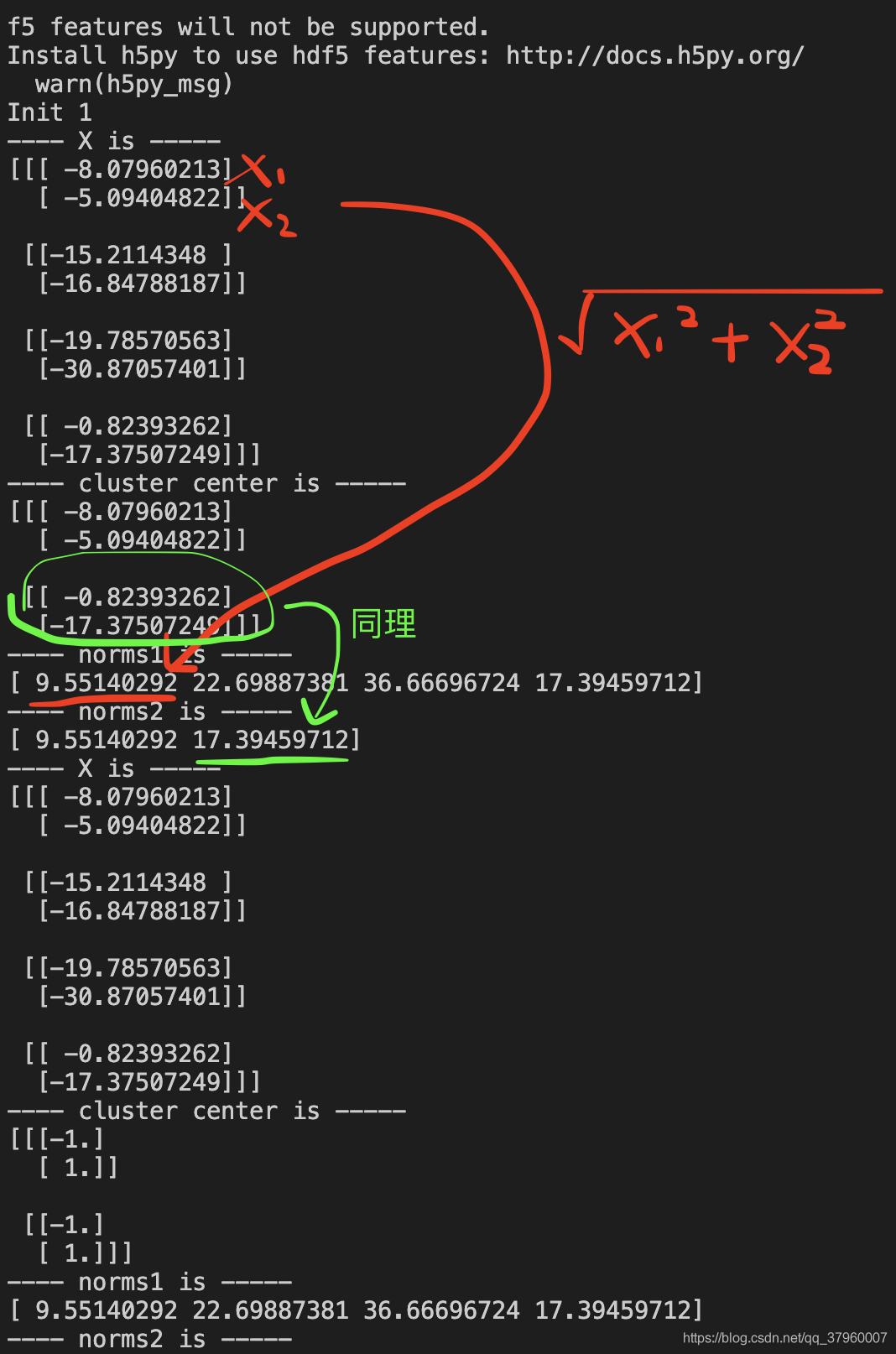
這驗證了norms確實是向量的模長,於是norms參數也解決了。至於self_similarity不動它,設定成False即可
測試
這裏有個坑,如果直接在你自己的py檔案中 import tslearn.cycc會報錯,可能是so檔案比較特殊。這裏採用了另一種方式進行測試,即再次直接修改原始碼,如下:
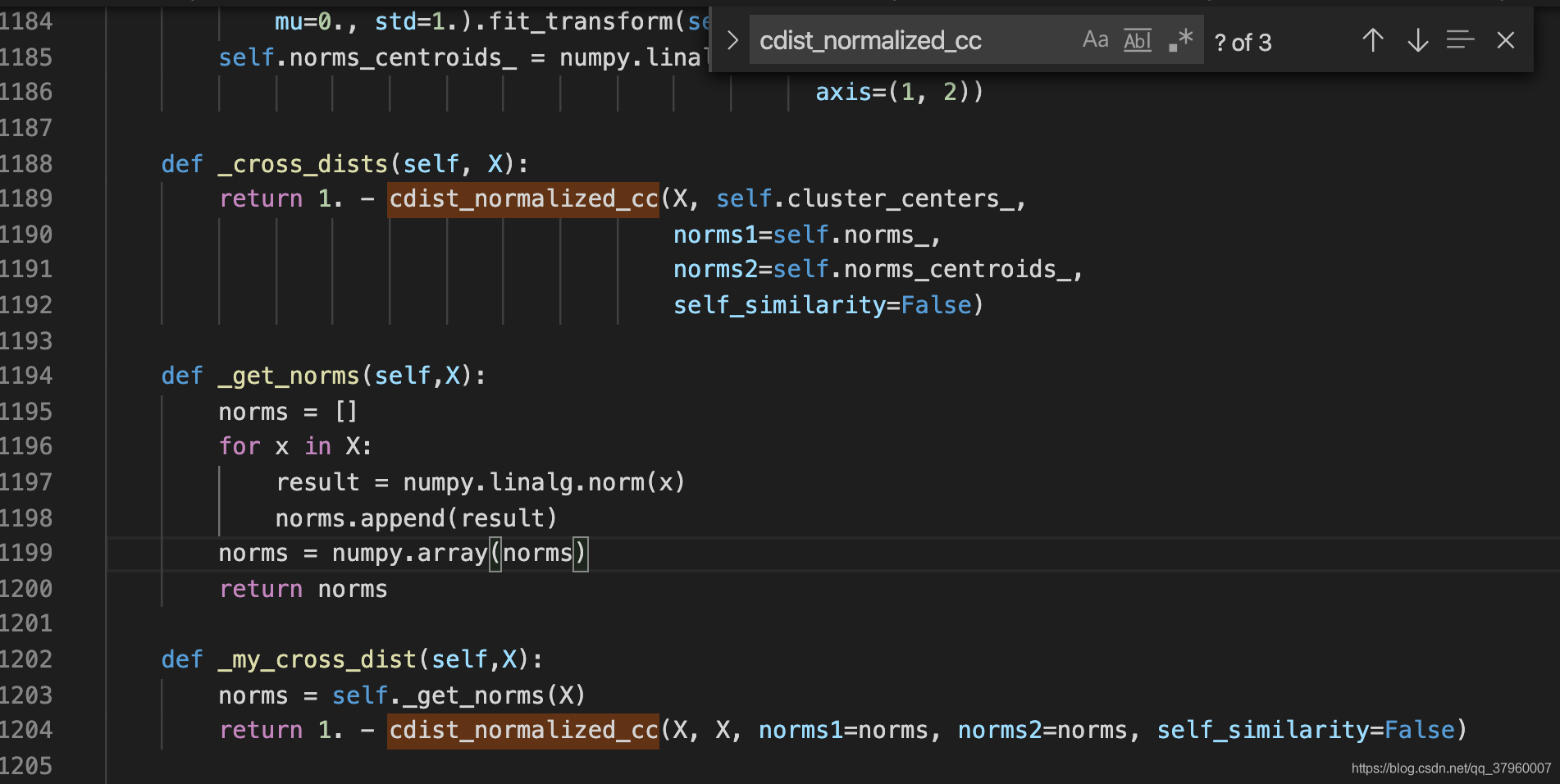
增加_get_norms函數和_my_cross_dist函數,前者負責計算模長,後者返回時序集合X的距離矩陣,即X中每個元素ti之間的距離所形成的矩陣。參數則按照前一小節的方式傳
下面 下麪先進行距離矩陣的測試
import numpy as np
from tslearn.clustering import KShape
from tslearn.generators import random_walks
def test_cross_dist():
seed = 0
num_cluster = 2
ks = KShape(n_clusters= num_cluster ,n_init= 5 ,verbose= True ,random_state=seed)
X = random_walks(n_ts=4, sz=2, d=1) * 10
dists = ks._my_cross_dist(X)
print(dists)
test_cross_dist()
隨機生成四條曲線,每條兩個時間點,呼叫_my_cross_dist測試,結果如下
[[ 6.68621811e-08 5.90374654e-01 3.93694605e-01 2.73371849e-01]
[ 5.90374654e-01 1.06040988e-08 1.06048454e+00 1.23859603e+00]
[ 3.93694605e-01 1.06048454e+00 -9.46784942e-08 9.19879423e-02]
[ 2.73371849e-01 1.23859603e+00 9.19879423e-02 -1.73142627e-08]]
可以看到對角線已經到了負8次方,幾乎是零了。距離矩陣測試成功
下面 下麪使用輪廓係數測試KShape聚類
import numpy as np
from tslearn.generators import random_walks
import tslearn.metrics as metrics
from tslearn.clustering import silhouette_score
def test_kshape_silhouette_score():
seed = 0
num_cluster = 2
ks = KShape(n_clusters= num_cluster ,n_init= 5 ,verbose= True ,random_state=seed)
X = random_walks(n_ts=20, sz=16, d=1) * 10
#隨機生成標籤(即亂猜聚類結果)
y_pred = np.random.randint(2, size=20)
dists = ks._my_cross_dist(X)
#這步重要,由於計算出的距離矩陣只是負8次方,還不是真正的零,得替換,否則會報錯
np.fill_diagonal(dists,0)
#計算輪廓係數
score = silhouette_score(dists,y_pred,metric="precomputed")
print(score)
在隨機設定標籤的情況下,理論上輪廓係數應該是零,表示一個元素既可以屬於這個簇,也可以屬於另一個簇,執行
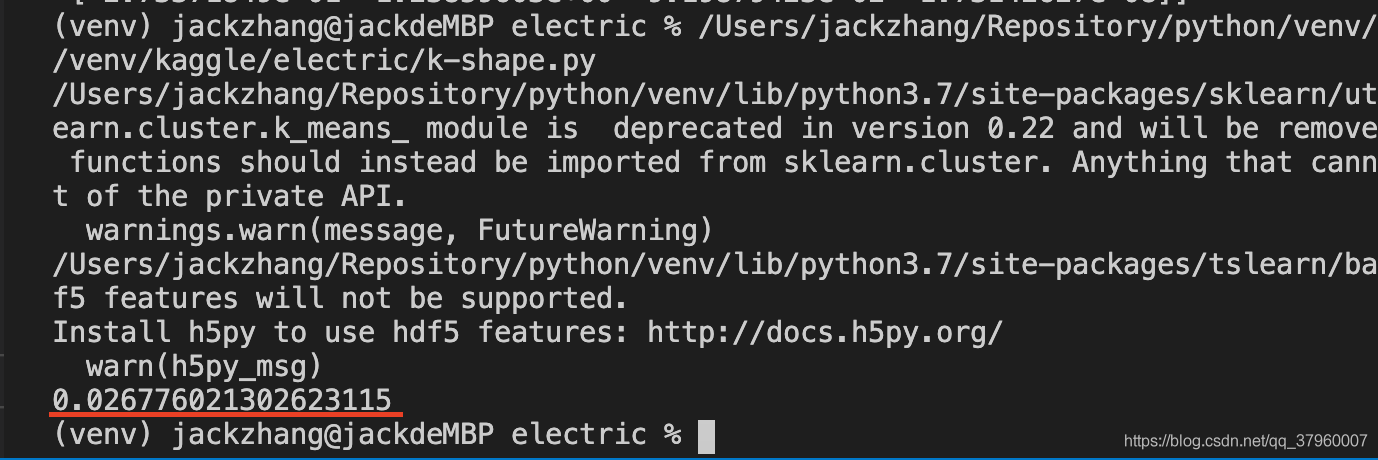
可以看到,輪廓係數十分接近零,測試成功
實戰
隨機情況下測試成功還不夠,下面 下麪使用KShape聚類測試真實生產數據,並使用輪廓係數進行評估
import numpy as np
import glob
from tslearn.clustering import KShape
from tslearn.preprocessing import TimeSeriesScalerMeanVariance
import matplotlib.pyplot as plt
from tslearn.generators import random_walks
import tslearn.metrics as metrics
#自定義數據處理
import data_process
# X,100個數據,每個2800+維
X = np.loadtxt("top100.txt",dtype=np.float,delimiter=",")
# 降維,2800維將成30維
X = data_process.downsample(X,30)
seed = 0
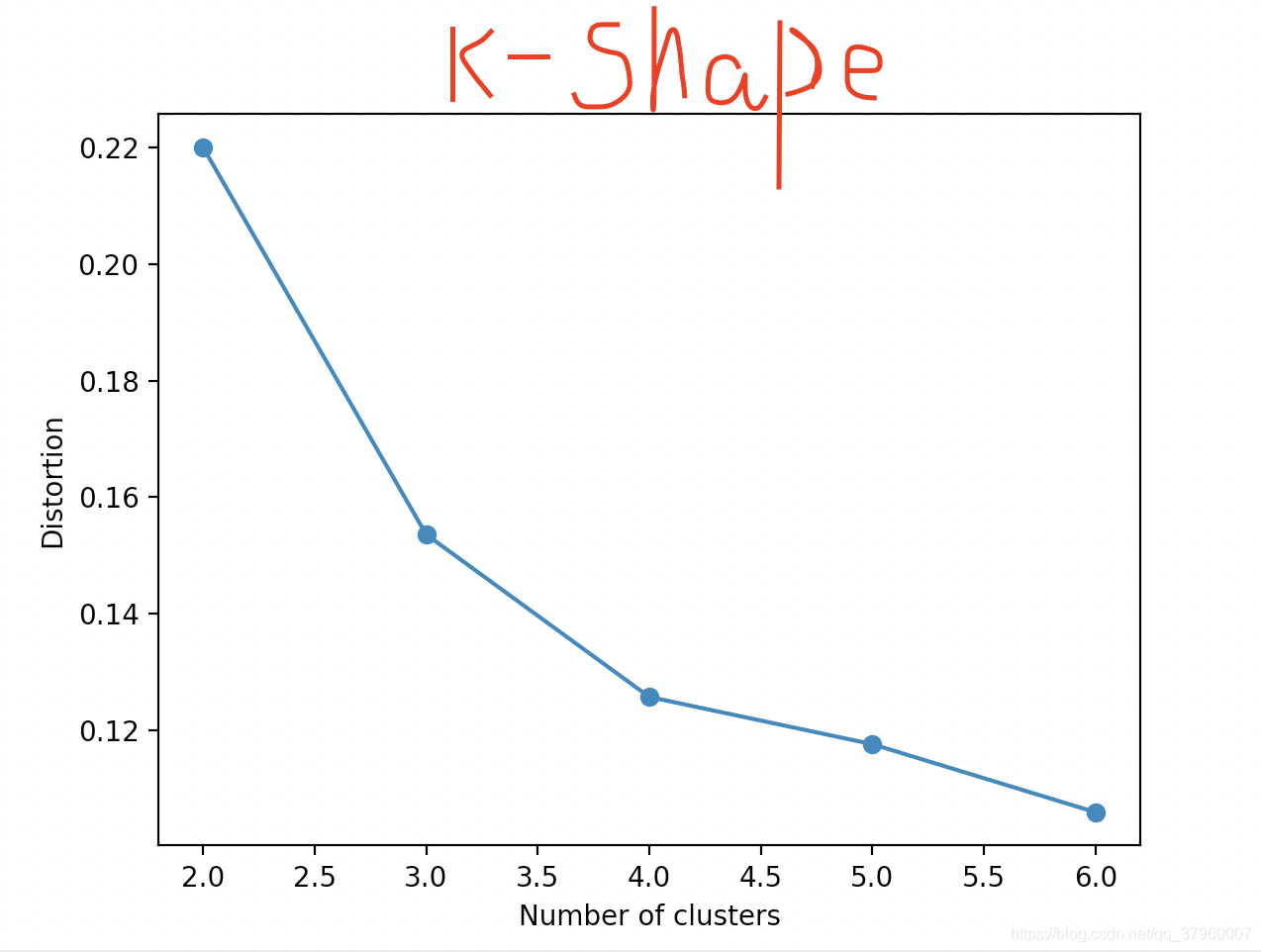
# elbow = 4(elbow法則尋找最佳聚類個數)
def test_elbow():
global X,seed
distortions = []
X = TimeSeriesScalerMeanVariance(mu= 0.0 ,std= 1.0 ).fit_transform(X)
#1 to 10 clusters
for i in range ( 2 , 7 ):
ks = KShape(n_clusters=i, n_init= 5 ,verbose= True ,random_state=seed)
# Perform clustering calculation
ks.fit(X)
#ks.fit will give you ks.inertia_ You can
#inertia_
distortions.append(ks.inertia_)
plt.plot(range ( 2 , 7 ), distortions, marker= 'o' )
plt.xlabel( 'Number of clusters' )
plt.ylabel( 'Distortion' )
plt.show()
def test_k_shape():
global X,seed
np.random.seed(seed)
num_cluster = 4
# 標準化數據
X = TimeSeriesScalerMeanVariance(mu= 0.0 ,std= 1.0 ).fit_transform(X)
ks = KShape(n_clusters= num_cluster ,n_init= 5 ,verbose= True ,random_state=seed)
y_pred = ks.fit_predict(X)
dists = ks._my_cross_dist(X)
np.fill_diagonal(dists,0)
score = silhouette_score(dists,y_pred,metric="precomputed")
print(X.shape)
print(y_pred.shape)
print("silhouette_score: " + str(score))
for yi in range(num_cluster):
plt.subplot(2, 2, yi + 1)
for xx in X[y_pred == yi]:
plt.plot(xx.ravel(), "k-", alpha=.3)
plt.plot(ks.cluster_centers_[yi].ravel(), "r-")
plt.text(0.55, 0.85,'Cluster %d' % (yi + 1),
transform=plt.gca().transAxes)
if yi == 1:
plt.title("SBD" + " $k$-shape")
plt.tight_layout()
plt.show()
elbow法則,4這裏有最大彎曲度,確定是4聚類
未降維之前的時序數據聚類效果:
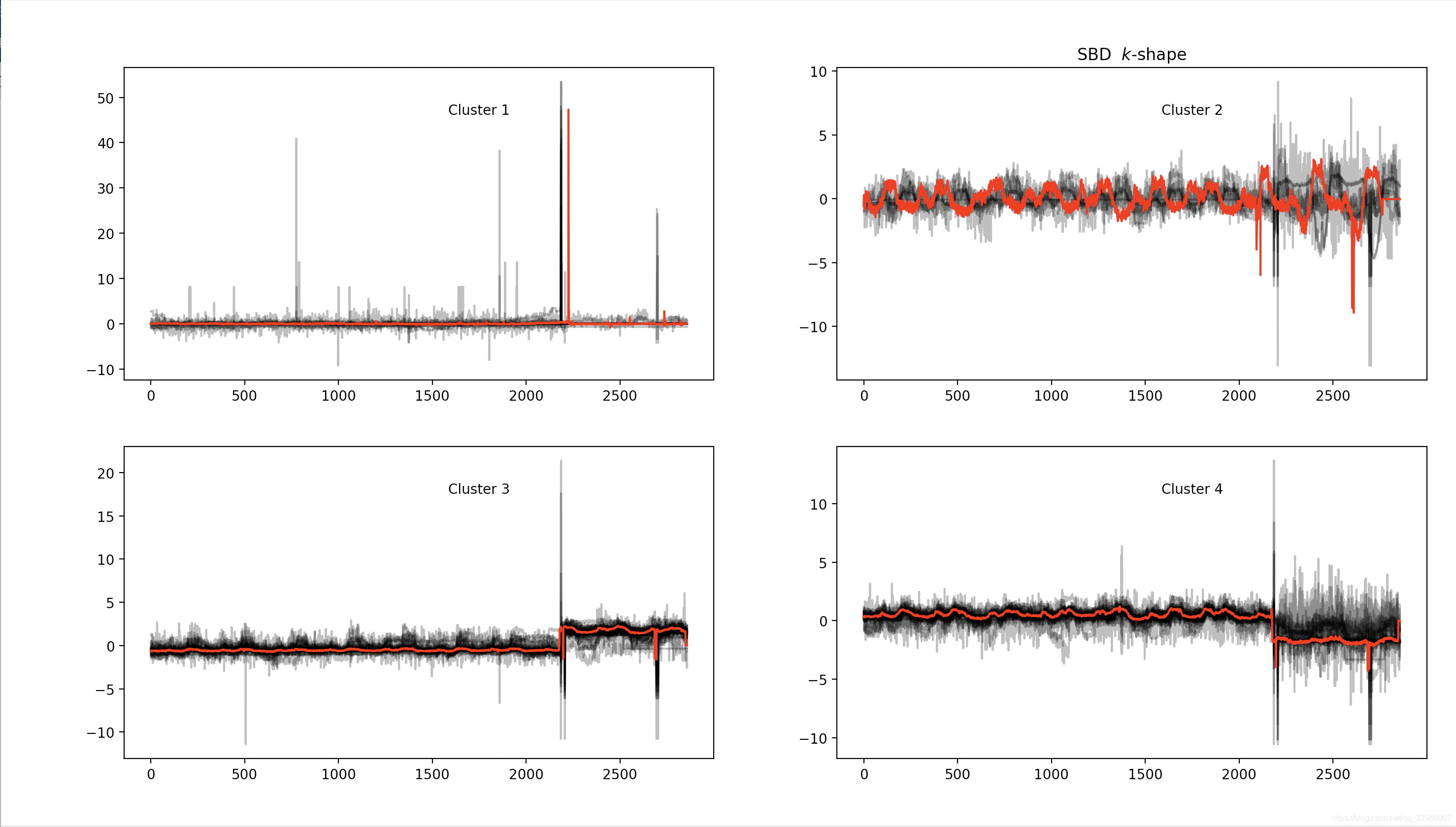
列印結果:
(100, 2858, 1)
(100,)
silhouette_score: 0.5171964754458871
降維之後的聚類效果:
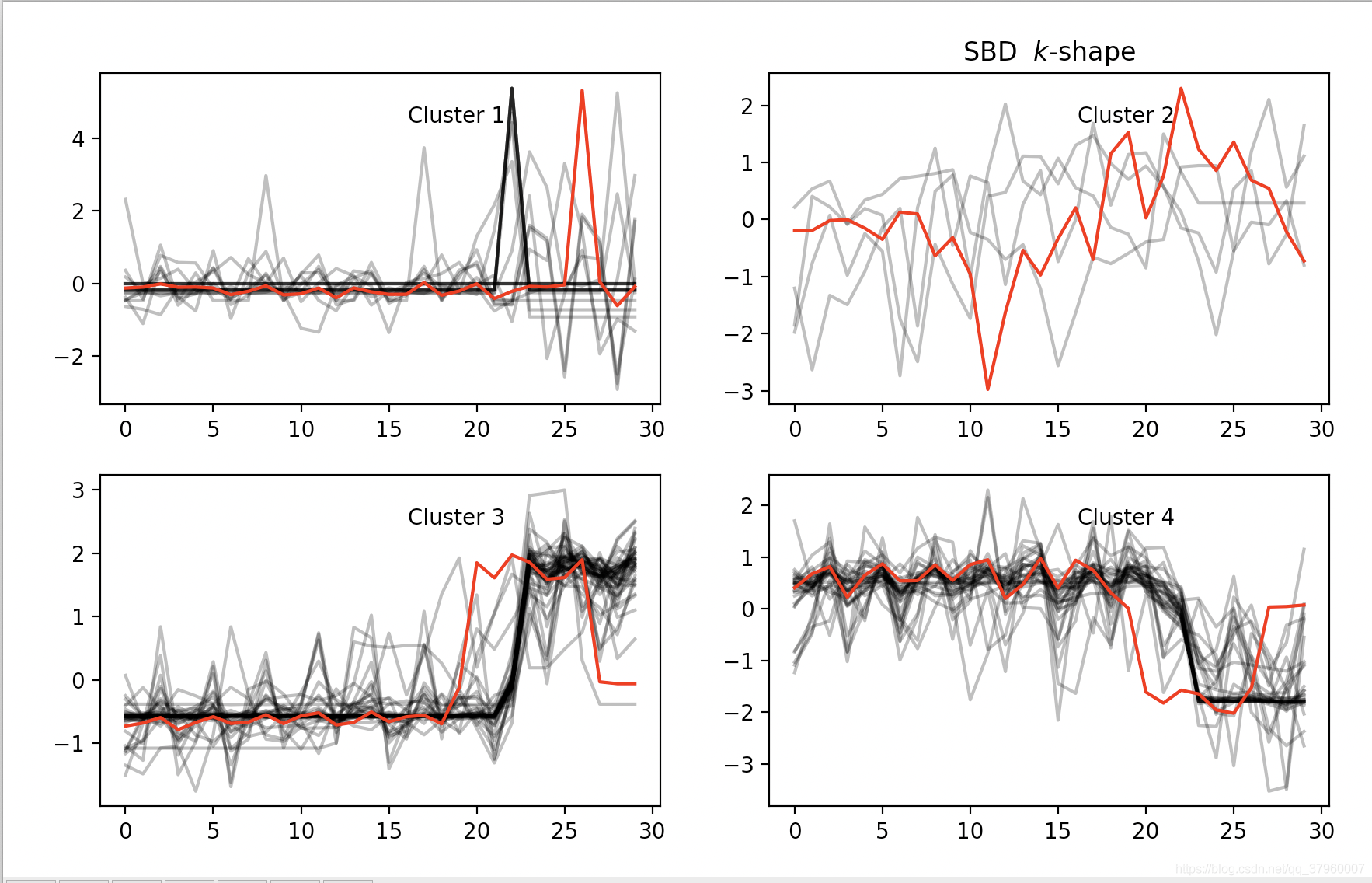
列印結果:
(100, 30, 1)
(100,)
silhouette_score: 0.5510349154221014
KShape聚類評價
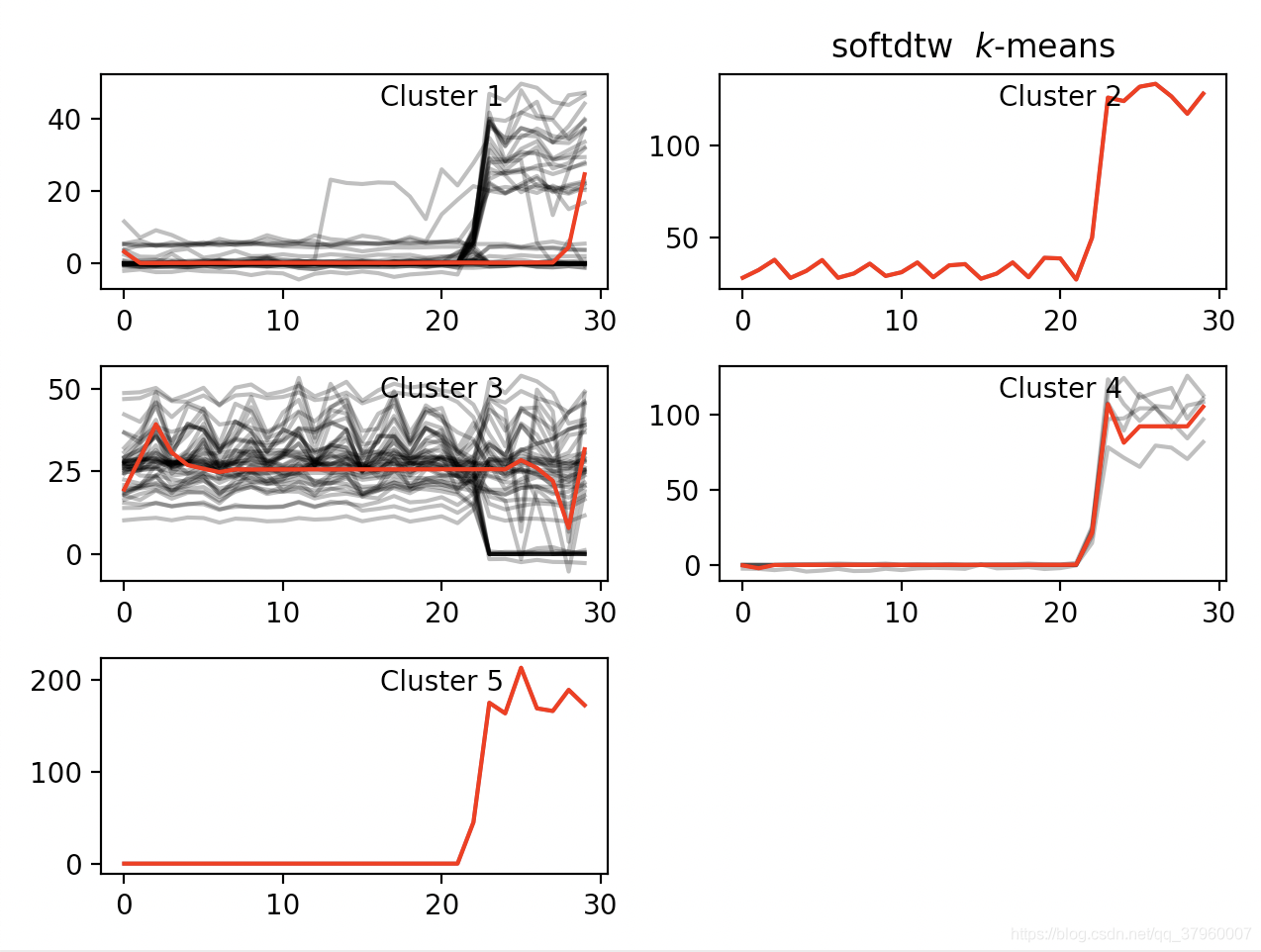
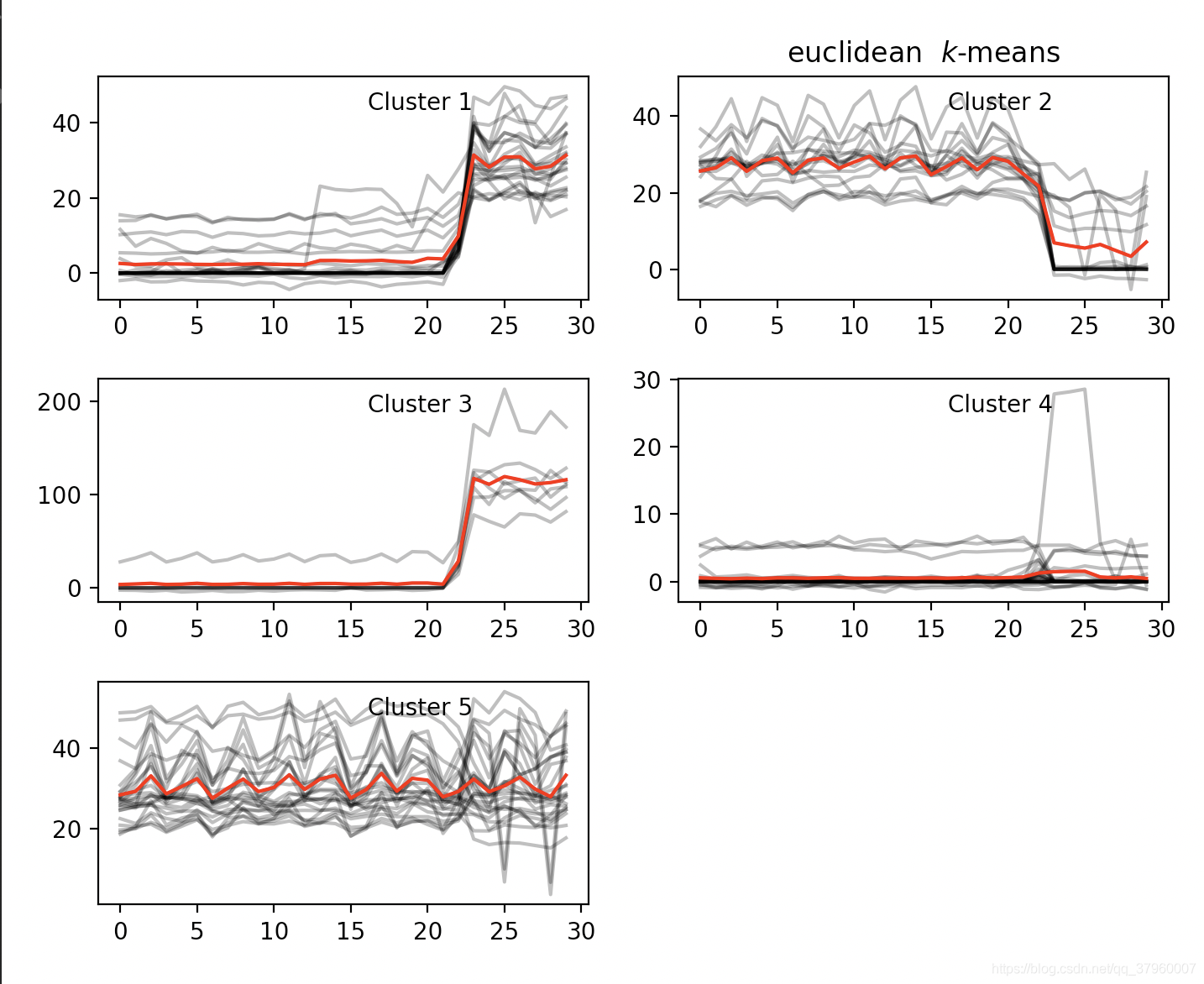
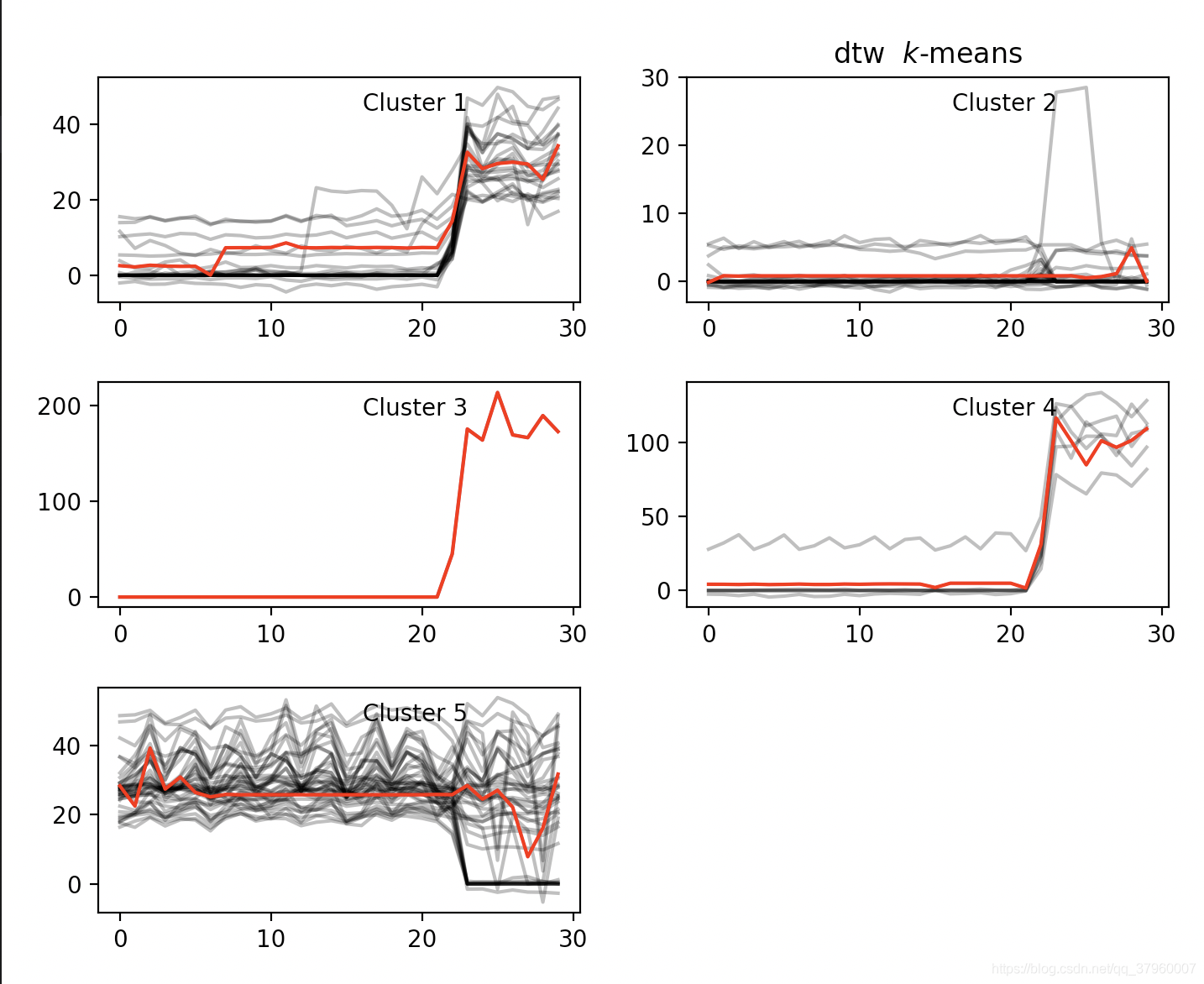
不同演算法的聚類效果圖對比纔是最好、最簡潔的評價,上圖
K-Means + 歐氏距離(同一數據集)# silhouette_score: 0.5095828273645443
K-Means + DTW + DBA # silhouette_score: 0.5878368758366767
K-Means + SoftDTW # silhouette_score: 0.7473192701567871